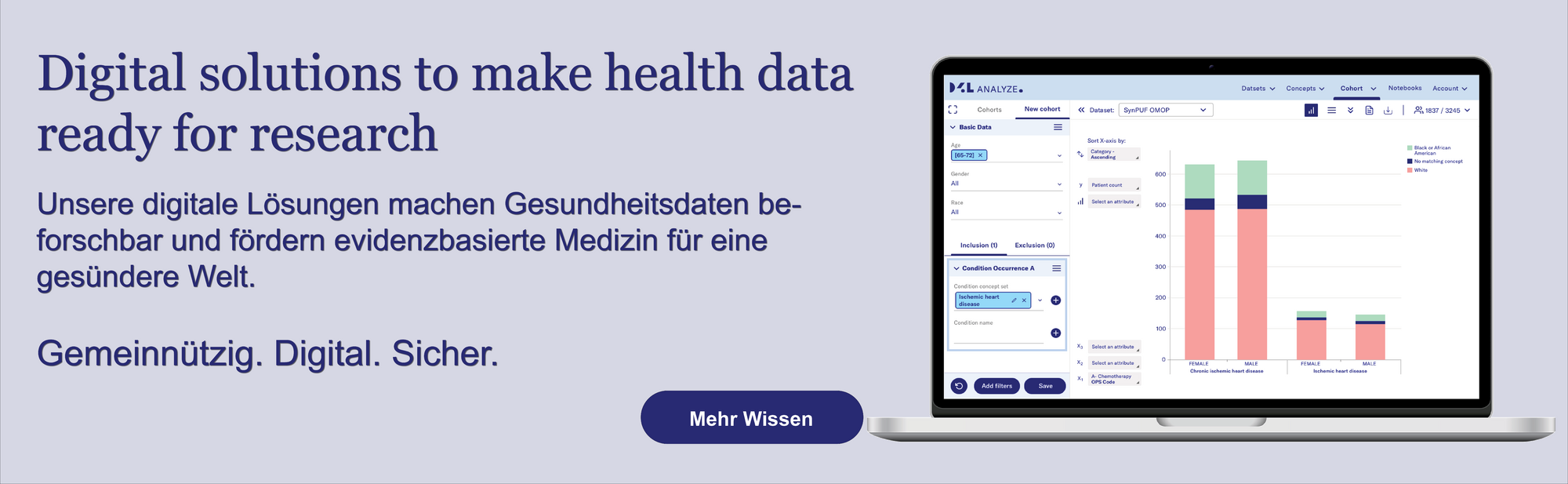
AlphaFold AI hat die Gesundheitsdaten Forschung in kurzer Zeit tiefgreifend verändert: Seit 2020 liefert die DeepMind-Technologie Proteinmodelle mit bisher unerreichter Präzision und macht sie der Community im Sinne von Open Science breit zugänglich – ein Meilenstein, der 2024 mit dem Chemie-Nobelpreis an Demis Hassabis, John Jumper und David Baker anerkannt wurde [1–3].
Was ist AlphaFold AI?
AlphaFold ist ein KI-basiertes Modell zur Proteinstrukturvorhersage. DeepMind stellte 2020 die Lösung des 50-jährigen „Protein-Folding-Problems“ vor und publizierte 2021 die methodische Grundlage (AlphaFold 2) in Nature. Parallel entstand mit EMBL-EBI die AlphaFold Protein Structure Database (AF-DB), die heute über 214 Mio. Einträge umfasst – nahezu das gesamte UniProt-Spektrum. Diese offene Ressource ermöglicht Gesundheitsdaten Forschung auf neuem Niveau, von Grundlagen bis Anwendung [1, 2, 4–6].
Open Science: AlphaFold-Modelle und die AF-DB sind frei zugänglich; die Daten (inkl. PAE-Plots) lassen sich programmgesteuert auswerten. Das beschleunigt Hypothesengenerierung in der biomedizinischen Forschung und schafft wiederverwendbare Assets für Gesundheitsdaten Forschung [5, 6].
Wie funktioniert AlphaFold AI?
AlphaFold kombiniert Deep-Learning-Architekturen mit Multiple Sequence Alignment (MSA), Template-Information und geometrischen Repräsentationen, um aus Aminosäuresequenzen 3D-Strukturen zu berechnen. Die Qualität bewertet das System u. a. mit pLDDT (lokale Vertrauensmaße) und PAE (Predicted Aligned Error) für domänenübergreifende Konfidenz – zwei Kennzahlen, die Forschenden helfen, Modellgrenzen korrekt zu interpretieren [2, 7–9].
AlphaFold 3 (2024): Das aktuelle Modell erweitert die Vorhersage auf Biomolekül-Interaktionen (Proteine, DNA/RNA, Liganden/Antikörper, Ionen) mittels diffusionsbasierter Architektur. Es verbessert damit die Aussagekraft für Bindungsszenarien, bleibt aber – wie jede In-silico-Methode – auf experimentelle Validierung angewiesen [3, 10].
AlphaFold AI in der wissenschaftlichen Entdeckung
In der Wirkstoffentwicklung verkürzt AlphaFold die Zeit von der Zielhypothese zur Struktur, erlaubt Priorisierung von Targets und unterstützt die Identifikation potenzieller Protein-Bindungsstellen. Reviews und Fallstudien dokumentieren raschere Hypothesengenerierung in Onkologie, Infektionsmedizin und seltenen Erkrankungen – ein klarer Mehrwert für Gesundheitsdaten Forschung [11–13]. Gleichzeitig bleiben Flexibilität, Allosterie und dynamische Taschen kritische Punkte, die experimentell zu prüfen sind [11].
AlphaFold 3 wird bereits in industrienahen Programmen eingesetzt (Isomorphic Labs; Kollaborationen u. a. mit Eli Lilly und Novartis), während die nicht-kommerzielle Forschung über den AlphaFold Server kostenfrei zugreift [14–17].
Anerkennungen und Auszeichnungen
Die Entwickler von DeepMind AlphaFold wurden 2024 mit dem Nobelpreis für Chemie ausgezeichnet (Hassabis; Jumper; Baker für computergestütztes Protein-Design) – ein Signal für die wissenschaftliche Tragweite und den nachhaltigen Impact auf die Strukturbiologie und angrenzende Felder [3, 18–20].
AlphaFold AI und die Zukunft der Systemmedizin
Mit AlphaFold 3, Omics-Integration (Genomik, Proteomik, Metabolomik) und digitalen Zwillingen wächst das Potenzial, Systembiologie datengestützt zu simulieren – vom Molekül bis zum Patienten. Entscheidend ist die Interoperabilität klinischer und molekularer Daten (z. B. via OMOP CDM, FAIR-Metadaten) und eine Governance im Einklang mit WHO-Leitlinien für Ethik und Regulierung von KI im Gesundheitswesen [6, 21, 22].
Die Inhalte dieses Artikels geben den aktuellen wissenschaftlichen Stand zum Zeitpunkt der Veröffentlichung wieder und wurden nach bestem Wissen und Gewissen verfasst. Dennoch kann der Artikel keine medizinische Beratung und Diagnose ersetzen. Bei Fragen wenden Sie sich an Ihren Allgemeinarzt.
Ursprünglich veröffentlicht am